Kristina S. R. Stenløkk kommer fra Larvik i Vestfold, hjem til Norges største bøkeskog. Hun var tidligere student ved BIAS, og er nå PhD-kandidat ved CIGENE, hvor hun studerer evolusjonen hos salmonider. Bioinformatikk er for henne et vindu inn til biologiens og evolusjonens verden – en verden som først fascinerte henne da hun som ung jente samlet biller i skogen med onkelen.
Kan du fortelle meg litt om deg selv?
Jeg begynte på NMBU i 2014, på linjen for Kjemi og Bioteknologi, retning molekylær-biologi. Bakgrunnen for det var at jeg var veldig interessert i biologi og evolusjon. På tredje året så skjønte jeg at bioinformatikk var min måte å komme nærmere evolusjon på, så da bytta jeg til retning bioinformatikk. Det å gå tilbake til og prøve å forstå historien til hvordan gener har blitt som de har blitt, det synes jeg er kjempespennende.
Hvorfor syntes du at akkurat bioinformatikk var veien inn?
Det er veldig mye bioinformatikk som må ligge til grunn for å kunne gjøre molekylærbiologi eller proteinkjemi. For ikke bare å gi bort analysene, så må man kunne stå i det selv.
Kan du fortelle meg litt om din ekstern masteroppgave fra Forsvarets Forskningsinstitutt (FFI)?
Jeg fikk først tilbud om en sommerjobb der, i 2018. Måten jeg kom i kontakt var at jeg sendte en åpen mail … så ble jeg invitert inn på et møte der, og da snakket de om at de testet ut hvordan man kan klassifisere det man finner i luft – virus, bakterier, sopp, pollen, osv. Så det fikk jeg lov til å jobbe med på sommerjobben, og på mastergraden. Grunnen til at dette er spennende er at det er gjort så lite forskning på luft som et biologisk habitat, og vi vet egentlig lite om hva som finnes der. Det er også viktig å forstå det biologiske innholdet i luft i sammenheng med sykdomsutbrudd.
Tok du luftprøver og analyserte hva som var i lufta rett og slett?
På FFI hadde jeg hele den pipelinen selv, da tok jeg ut og satte opp et forsøk hvor jeg samlet inn luft. Det er ganske fascinerende – det er en sånn støvsuger, hvor du setter et filter på, og så trekker du masse luft gjennom det filteret. Så kan du DNA-isolere det filteret, så sekvenserer du det, og så kan du bruke ulike bioinformatikkverktøy til å se hva du egentlig har da.
Det store jeg fant var at det var veldig dårlige databaser for luft. Optimalt da, så hadde det vært en prøve på alle organismer man kan finne som lå i den databasen, men det var opptil sytti prosent av det jeg fant som ikke matchet mot noenting. Så det betyr enten at det er for dårlige sekvenser, eller at det er ikke kartlagt godt nok.
Hvordan var det å være student ved BIAS egentlig?
Det var kjempebra! For det første så var det utrolig bra oppfølging av Lars Snipen (veilederen). Så kjente jo jeg mange av de på BIAS fra før for jeg har vært hjelpelærer i bioinformatikk, BIN210, og i STIN100. Det er jo et relativt lite miljø, så man blir veldig godt kjent med folk veldig fort. Så fikk jeg lov til å være med på de BIAS møtene – og følte det var veldig hyggelig å få være med på og spise lunsj etterpå og bli kjent. Det tror jeg er en ganske stor fordel, sammenliknet med mange andre grupper, det at det er såpass lite og trygt og vennlig. Man føler seg veldig godt tatt imot.

Hva gjør du nå på CIGENE – fra luft til laks?
Det er et ganske stort hopp, men det er fortsatt bioinformatikk. Det er mer mot evolusjonsbiologi. Laksefamilien, eller salmonider, har vært gjennom en hel-genom duplisering, som er så nylig at man fortsatt kan prøve å studere effektene av det. For etter at et genom har blitt hel-genom duplisert, så er det stort press på evolusjon for å komme tilbake til den stabile formen: diploid, altså to kopier av genomet. Da skal jeg se både spesifikt på atlantisk laks, og sammenlikne med flere arter i salmonidene.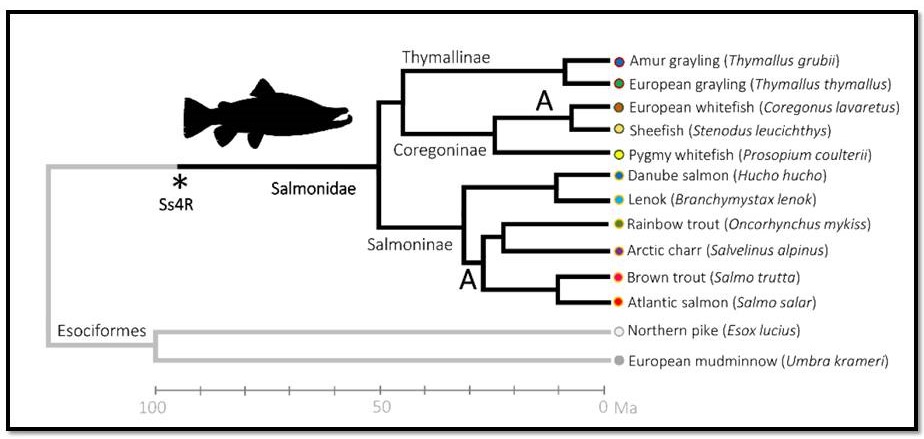
Det prosjektet jeg jobber i heter TRANSPOSE. Det jeg skal se på nå er hvilke mekanismer har vært med i den rediploidiseringen, altså tilbake til to kopier. Jeg ser på transposoner, som er DNA-sekvenser som flytter seg rundt i DNAet, siden vi tror at transposoner er knyttet til evolusjonen. Det har vært vanskelig å studere variasjon i genomet på grunn av transposoner, siden disse elementene ofte er mye lengere enn sekvensavlesningene man får fra teknologien som har vært brukt. Med ny teknologi kan man studere dette på en helt ny måte. Å forstå hvordan transposoner påvirker evolusjon er vel det ultimate målet.
Hvordan ble overgangen da, fra masterstudent til arbeidslivet nå i dag?
Jeg er ganske fersk, men det har vært veldig bra at jeg har fått bli med på et prosjekt som allerede er i gang, og får være med på ting som ikke har blitt gjort før. De sekvenseringene her er helt nye og banebrytende, og så får jeg lov til å få masse ansvar med en gang. Så uten tvil, bratt læringskurve, men også ekstremt givende, da. En ting jeg har merket er at det er annerledes å være ansatt enn når man er student. Man er mye friere og har kanskje litt mer å slå i bordet med. Det føles veldig som jeg er en del av noe nyttig da.
Hvorfor tok du valget om å begynne som stipendiat?
For meg, siden jeg gikk på barneskolen, så har jeg vært sikker på at det er forskning jeg vil gå mot. Siden jeg var liten så har jeg hatt en onkel som har hatt veldig stor innflytelse på meg. Han er veldig opptatt av natur og samling, så vi har alltid vært ute og samlet insekter og sånt. Å se hvor forskjellig ulike billearter kan være – i form, farge og funksjon – har vært en ekstrem fascinasjon siden jeg var liten.
Så føler jeg også at bioinformatikk er veldig forskningsrettet sånn det er nå da. For å jobbe som forsker "kreves" det ofte doktorgrad. Sånn det er nå så er det ofte enten på et sykehus eller akademia som man fortsetter. For meg så dukka den stillinga her opp, og da var det på en måte ingen tvil i det hele tatt – det var perfekt.
Hvordan gjorde masteroppgaven din deg rustet for arbeidslivet?
Det som på en måte var en sjokkopplevelse – en stor lærdom fra masteren – var at når du kommer forbi det å ta fag, så er det ingen som har fasiten. Du må bare finne ut av det selv, og det er ingen som er ekspert på akkurat det du prøver å finne ut, så det føler jeg masteren var en god forberedelse på; forskning er veldig mye problemløsning og uoppgåtte stier, for å si det sånn.
Er det noe råd eller tips som du kan gi til en student nå om studieretning eller masterprogram når de skal tenke på karriere etterpå?
Det mange kanskje ikke tenker på er at valg av veileder er vel så viktig som valg av tema på masteroppgaven. Det er gøy å ha en veldig spennende oppgave … men det kan være ekstremt demotiverende å sitte der uten å føle at du får den hjelpen du trenger. Så det med en god veileder det er alfa og omega. Så er det en veldig god idé å prøve å bli litt kjent med mulige veiledere, sette i gang tidlig og prate med folk for å høre om mulige masteroppgaver.
Med tanke på karriere videre er det så klart lurt å følge med tidlig. Man vet jo aldri hva som ligger klart når man skal søke selv, men å ha et visst inntrykk av hva som pleier å komme, da, det er lurt.
Har du tenkt noe på mulighetene du kommer til å ha etter stipendiatet?
Det er jo uten tvil en karrierevei hvor det er vanskelig å ha en fast jobb. Men jeg har inntrykk av at det er større og større behov for bioinformatikere, så jeg håper at det dukker opp et eller annet i forskningstreet.
Noe jeg føler er spesielt med bioinformatikk er at man fremdeles ikke er helt ferdig "utdannet" etter et master. Bioinformatikk handler veldig mye om "learning by doing" - og krever mer enn teoretiske fag. Man må rett og slett jobbe seg igjennom masse problemstillinger for å bli god.
